r实战系统发育树,系统发育树构建与可视化技巧解析
时间:2025-01-21 来源:网络 人气:
探索进化奥秘:R语言实战构建系统发育树
想象你手中握着一把打开生物进化奥秘之门的钥匙。这把钥匙,就是R语言,一种强大的编程工具,它能帮助你构建出揭示物种间演化关系的系统发育树。今天,就让我们一起踏上这场探索之旅,用R语言揭开生物进化的神秘面纱。
一、系统发育树的魅力
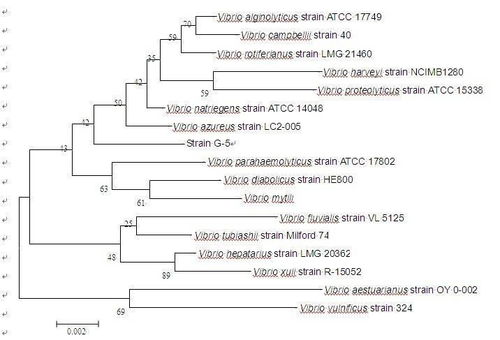
系统发育树,又称进化树,是生物学研究中不可或缺的工具。它通过展示物种间的遗传相似性和差异性,帮助我们理解生物的演化历程。想象一棵树上的每一片叶子都代表一个物种,而树枝则象征着它们之间的亲缘关系。通过这棵树,我们可以清晰地看到物种是如何从共同的祖先演化而来的。
二、R语言:构建系统发育树的利器

R语言,作为一款功能强大的统计软件,在生物信息学领域有着广泛的应用。它不仅可以帮助我们进行数据分析和可视化,还能轻松构建系统发育树。下面,就让我们一起来学习如何使用R语言构建系统发育树。
三、R语言实战:构建系统发育树的步骤
1. 数据准备:首先,你需要收集或获取物种的DNA或蛋白质序列数据。这些数据通常以fasta格式存储。接下来,使用R语言的Bioconductor包中的read.fasta函数读取序列数据。
2. 序列比对:为了比较不同物种之间的序列相似性,我们需要进行序列比对。R语言中,常用的序列比对工具包括Clustal Omega和MUSCLE。使用Bioconductor包中的ClustalW或MUSCLE函数进行序列比对。
3. 构建系统发育树:完成序列比对后,我们可以使用R语言的Phylogenetics包中的函数构建系统发育树。常用的建树方法包括邻接法(Neighbor-Joining)、最大似然法(Maximum Likelihood)和贝叶斯法(Bayesian Inference)。
4. 树状图可视化:构建好系统发育树后,使用R语言的ggtree包或ape包将其可视化。这些包提供了丰富的绘图选项,可以帮助我们更好地展示树状图。
四、案例分析:使用R语言构建青鳉属系统发育树
以青鳉属为例,我们可以通过以下步骤使用R语言构建其系统发育树:
1. 数据准备:从NCBI数据库下载青鳉属物种的16S rRNA基因序列。
2. 序列比对:使用Clustal Omega进行序列比对。
3. 构建系统发育树:使用Phylogenetics包中的函数,采用最大似然法构建系统发育树。
4. 树状图可视化:使用ggtree包将构建好的系统发育树可视化。
通过以上步骤,我们可以清晰地看到青鳉属物种之间的亲缘关系,从而更好地理解其演化历程。
五、:R语言在系统发育树构建中的应用
R语言在系统发育树构建中具有广泛的应用。它不仅可以帮助我们进行数据分析和可视化,还能提供多种建树方法和绘图选项。通过学习R语言,我们可以轻松构建出揭示生物进化奥秘的系统发育树。
在这个充满挑战和机遇的时代,让我们用R语言开启探索生物进化奥秘的大门,共同见证生命的奇迹!
教程资讯
教程资讯排行